阅读:0
听报道
导语
针对复杂网络的分析方法琳琅满目,然而对于网络科学初学者,或者涉及网络的跨学科研究者而言,要快速理解大量相关概念和方法颇有困难。在发表于的一篇预印本论文中,作者以生物学中的各类为例,提供了9个入门网络数据分析的技巧,帮助初学者避免常见的陷阱。
从分子到生态系统层面,生物系统通常可以被表示成一系列相互作用的实体。近期在数据采集技术上(例如:高通量的检测和记录)取得的大量进展提供了一个量化这些相互作用的机会,也因此为处理这些数据的方法提出了更高的要求。在此背景下网络被广泛地应用在生物学、生物信息学、生态学、神经系统科学和流行病学以来表征相互关联的数据。一个网络包含一组实体(节点或顶点),这些实体通过描绘某些交互或关系的边(或链接)连接起来。这些关系通常是在元数据中被直接观察或者从中推断得出的。
例如第一个例子包含了一个蛋白质-蛋白质相互作用网络(PPI),其中两个蛋白质之间的互动通过实验估算得到,还有在田野间直接观察到的植物和传粉者之间的相互作用。从基因表达的数据重建基因调控网络,从物种丰度推断出共现网络或者从GPS轨迹推导出不同动物间的社交网络是第二种情况的一些例子。新的网络类型(例如,细胞-细胞相似性网络[2],Hi-C网络和图像相似性网络[3])也一直不断地出现。
网络是非常吸引人的事物,研究人员已经开发了许多方法用来分析它们的结构。然而,在生物学方面的网络一般不是由相关专家进行分析的,对于他们来讲,理解大量相关领域的概念和可用的方法往往是困难的。在本文中,我们提供了九个技巧,以避免常见的陷阱,并增强生物学家对网络数据的分析能力。
技巧一:先阐述清楚问题,再使用网络分析工具
网络科学理论已经发展成熟并且功能强大,但是不能被用作“黑箱”。实际上,建立网络本身不应被视为目的。我们建议:1)在操纵数据之前首先列出科学的问题和假设列表。2)再进一步评估这些问题是否可以自然的转化为用网络分析的问题。而不是首先运用网络分析然后检查是否存在问题(与文献[4]中的规则1一致)。实际上,用网络表示和建模数据可以立即完成,但是要从基于网络的分析中提出问题就要困难很多。
为此,除了要集成网络的形式之外,更重要的是用网络来看问题的视角。它依赖于一种基础假设,这种假设既是网络建模力量的来源,也面临相应的挑战:任何发生的或者没有发生的相互作用,都必须在其所处背景环境中被分析。按照这种观点,任何节点对(A、B)之间的互动都应该被考虑进其它包含节点A或者B的节点对的环境。例如,两个基因之间的特定边的重要性取决于节点上对应的基因是否是一个中心基因(hub gene)(由许多其他基因控制)。这个视角不认为互动是独立的,因此与逐个考察每个交互的集合这样的方法完全相反。
最后,在进行任何数据分析之前,最好先检查你的问题和数据是否真的适合网络的视角来分析。如果节点或者边很少,虽然可以进行网络分析,但是结果可能会令人失望,因为没有足够的相互作用数据来识别数据中的结构。而另一方面,尽管可以将许多矩阵视作网络(每个格子连接一条边,请参见下一个技巧),但考虑使用专用于完整矩阵的非网络方法通常更为合适。例如,可以自然地用分层聚类或主成分分析来分析可能被视为相关网络的相关矩阵。换句话讲,当分析一个数据矩阵的时候,网络分析并不一定是适合的方法。
技巧二:正确地分类你的网络数据
要掌握网络领域的前沿概念和方法,了解图论中的术语是先决条件[5]。通常,适当地分类你的网络很重要,这样可以保证使用合适的方法。不同类型的网络适合不同的数据同样适用不同的方法。
连接可以是有向的(从源节点指向目的节点),这其中可能包括自指环(例如,与自身相互作用的蛋白质或食物网中的同类相食)。为简单起见而忽略这个信息实际上会背离原始数据。当处理具有嵌入值(权重)的边时,我们强烈建议你避免使用任何临时阈值将网络简化为二值化网络( Binarized network )。实际上,它清除了可用信息的重要部分,因为在二值化网络中某些方面的结构可能无法检测出来 [6]。二值化只能用作探索阶段(例如,为了方便对网络进行初始的可视化-参见技巧4),但它可能会使你的分析产生偏差(例如,嵌套模式可以在二值化生态网络中观察到,但不能在加权网络中观察到 [7])。通常存在一些可用于处理加权网络的方法,使得分析更有效。此外,数据分析者必须非常谨慎,因为在研究中,权重可以被认为是基于强度的(权重越大,连边越强)以及基于距离的(权重越小,节点越近)。
节点可以属于不同的类别,并且存在仅有可能在不同类别的节点之间的边(bi / tri / multi-partite网络;例如,节点作为宿主/寄生虫或作为植物/真菌/种子传播网络[8])。处理这些特殊网络,需要特定的方法。例如,许多统计方法依赖于预期的连接数量(例如,在关于网络模块的计算中,参见技巧5),与一元网络的处理相比,这些网络的处理方法不同。
最后,节点通常包含其他信息。例如,节点可以具有空间位置(例如,节点作为二维中的小块栖息地或农场,三维中的脑区域)或者可以与外部属性(例如,食物网中的物种特征)相关联。可以在分析中显式地考虑这些额外的信息,以了解它是否有助于组织网络[9],或者搜寻那些一旦考虑到其影响就会产生的额外结构(例如,空间的[10]或系统生成的影响[11])。在前一种情况下,一种更简单但次优的替代方法通常是使用后验信息来解释结果(例如,解释具有空间信息的基因网络的结构[12]或比较网络的结构与网络的元数据[13])。
技巧三:使用特定的网络分析软件
已经有一系列各种各样的软件可用于网络分析。尝试使用不具有针对性的工具就是浪费时间。这些软件工具属于两个截然不同的类别:基于图形用户界面(使用鼠标导航)和基于软件包(命令行界面或编程)。第一类主要致力于强大的交互式可视化(见技巧4)。它包括两个主要的开源软件工具Gephi和Cytoscape,两者都有活跃的社区作为支持。它们还提供了对某些网络指标的计算(在技巧5中讨论了相关指标的选择)。第二类主要是两个领先的通用网络包NetworkX和igraph,但是也存在大量更专用的包(例如R中的bipartite)。最近,基于浏览器进行的可视化[14]成为一个中间类别,主要基于一组javascript库(例如,Sigma.js)。
也就是说,我们强烈建议你学习编程并编写分析脚本(与“10个简单规则”中关于计算技能和可重复性的观点一致[15,16])。编写可复现的代码可以增强网络研究:在不同的数据集上,你可以毫不费力地重新运行对原始数据的修改版本的完整分析,并与对建模方法感兴趣的其他同事共享代码。最后,存在一组有限的常见网络文件格式(例如,用以描述邻接边的格式:邻接列表),你应该从一开始就采用这种格式,以便在不同的软件工具之间轻松切换。
同时,数据分析者应该避免仓促地使用这些工具中实现的不同函数。如技巧5和6中所强调的那样,至关重要的是在运行函数之前理解度量/方法以及根据问题和手头的数据选择合适的度量/方法。
技巧四:请注意,网络可视化可能很有用,但可能会产生误导
网络的一个强大方面是它们能够在单个视图中描绘复杂数据。因此,在二维平面上以图形方式展示网络是很吸引人的:节点分布在平面中,用边连接节点以实现最美观的展示(节点的定位称为布局)。这个看似简单的任务实际上是一个非常困难的组合优化问题。尽管可用网络的规模不断扩大,仍然有一个活跃的研究团体提出一系列启发式方法,旨在在合理的时间内获得良好的网络视图。上述提到的工具(见技巧3)中集成了各种易于使用的布局。
图形通常被认为是探索数据分析的重要工具[17]。但是,需要特别注意不要过度用网络可视化解释。布局不止提供了使网络好看的可视化,还能优化你经常忽略的给定指标(例如,最大化连接节点之间的吸引力)。也因此,使用眼睛看到的东西可能会存在偏差。请始终牢记,在进行网络可视化时,显示出来的节点的位置不是数据的一部分,而是来自算法处理的结果。因此,两个节点之间的距离不应被解释为接近程度的内在度量,因为另一个显示算法将可能产生不同的距离(参见图1a-b中两个红色节点之间的距离)。另一方面,网络可视化可用作说明网络分析结果(如技巧5和6中所示)。在这种情况下,应选择适当地布局,以突出显示网络属性(图1c)或由分析得出的结论(图1d)。例如,可以根据一些特定感兴趣的度量值来定位节点[18]。无论如何,我们鼓励生物学家清楚地描述科学出版物中网络的任何可视化中使用的布局,尤其是使其具有可重现性。
最后,我们还建议考虑将邻接矩阵可视化为热图/有色矩阵(参见[19]中对图1进行的解释)。该方法的优点在于能表示边的存在/权重,也能突出边的缺失。当矩阵行/列以信息方式重新排序时尤其相关(例如,在根据某些度量值进行排序时[20]或根据某些聚类结果排列时;参见提示5和6以及图1d)。
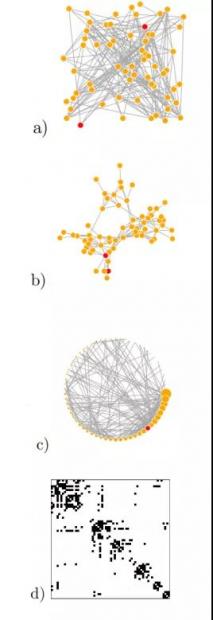
图2:四种对64位社会纺织者之间的互动建模网络的可视化图形。
图2:四种对64位社会纺织者之间的互动建模网络的可视化图形。a)随机布局。b)Fruchter and Reingold 布局。c)节点的大小和位置由其节点度决定的圆形布局。在面板a-c中,所展示的节点使用红色标记,可以看出它们的距离根据布局的变化而变化。d)使用Infomap算法聚类后的按行排列的邻接矩阵表示法(详见[22])。以上使用igraph进行网络可视化处理。
技巧五:避免盲目使用指标,而是要理解公式
由于网络可视化的局限之处,计算统计学摘要也可以(可能更好地)描述网络。初学者可以很快地发现一系列的网络度量:针对每个节点或者边的指标(局部指标:例如度)或者针对整个网络的指标(全局指标:连通度/密度或者模块性)。现在已经产生了大量的度量,我们强烈地建议你花时间仔细地阅读用到度量的数学定义(参见技巧9):数学理解越深,解释就越容易。例如:节点中心性概念可以通过一系列的度量来进行衡量,然而这些度量之间却各有不同。
此外,使用上述软件工具计算任何指标都非常容易,有时会妨碍分析者检查这些指标各自的优缺点。举个例子:当读到广泛使用的中介中心性(betweenness centrality)的定义,你可以理解它是基于最短路径的。如果你打算使用此度量,则有必要检查最短路径是否是与所研究过程中涉及的概念(例如食物网中的能量通量)相关或者是否存在问题(例如,功能网络中的路径实际上可能并不对应于信息流[19],当研究的问题并不是信息或疾病的扩散时,相关连接网络中的路径可能并不相关[23])。另一个例子是在有向/加权图的分析ß中引入不同的衡量指标。需要注意的是,加权度的公式要考虑两个方面的影响:有多少个邻居节点和它们权重有多大,这两个影响是不可能分离的(加权度2可以对应于权重2的一条边或权重0.5的四条边)。对于加权路径(在[24]中突出显示的潜在陷阱)也有类似的问题。最后,通常使用全局度量来比较网络(从不同数据或条件测量出来的网络,或者如技巧7所述的模拟出来的网络)。
在这种情况下,比较值时应特别小心,因为度量差异可能是简单的网络特征差异导致的副作用,比如节点或边的数量(参见[25]中提到的大脑网络分析中常见的疏漏和关于生态网络的指标和特征的共同变化的讨论[26])。事实上,模块化,模块数量和网络规模之间会相互影响[27]。
作者不是选择适合特定问题的特定度量,而是可能计算了所有可用度量中的大部分。当然,许多指标是相关的(参见[23]中的相关研究),因此有必要处理这种冗余度量以便于解释结果(例如,使用排序方法[28])。这种方法不是由假设驱动的(如技巧1所指出的),毫无疑问,它可以被每次选择一个度量来检查与数据的基本问题相关联的特定假设的增量方法所取代(对于其他一些统计分析,参见[4]中的规则5)。
技巧六:避免盲目使用聚类算法,最好检查它们之间的差异
随着近十年来数据爆发式增长,导致网络规模越来越大,聚类已经成为理解网络结构最流行的工具之一。其目的是将节点聚集成簇,以识别网络中的中尺度结构(即缩小网络)。选择网络聚类方法与选择网络度量会产生相似的问题(参见技巧5)。它不仅仅是使用软件中可用的一个功能。事实上,类似于任何其它的聚类方法,那些专门用于网络的方法都是为了汇集相似的对象(即节点),因此依赖于特定的节点相似性的定义。那么分析者会希望什么在网络中有相似之处呢?讨论不同方法的优缺点超出了本文的范围,然而已经有大量关于这一主题的文献(见技巧9)。下面,我们用三个经典的(相较于其它的方法)方法来说明选择一个特定的节点相似性的定义带来的影响。
节点相似性的第一个自然定义是它们之间存在一个连接。基于这个定义,网络聚类包括寻找模块化结构,即识别与其他节点间稀疏连接的密集节点群(也称为模块或社区)。社区检测法[22]实现了这种功能,它隐式地假设网络中存在模块。它们已经被成功地应用于生物学的许多研究中(例如识别染色质结构域[29])。第二种方法认为,当两个节点倾向于与相同类型的节点连接(或断开)时,它们是相似的。因此,如果食物网中的物种有相似的猎物和捕食者时,它们就被认为是相似的[30]。这个定义可以适应非模块化的网络结构[31],因为它假设涉及到的节点处于“多样的中尺度结构(meso-scale architectures)”中[32]。
随机区块模型(stochastic block model SBM)是基于这一定义的一种流行的方法[31,33],它已被应用在与一些生物网络相关的分析中(以突出生态网络中连接体[32]或功能群的复杂结构[34])。其中一个重要的特征是它允许通过不同的统计分布明确地模拟边的方向和权重[11]。第三种方法考虑对每个节点关联一个特征向量,然后把相似特征的节点聚集在一起。这包括基于模式(motif)的方法[35]和大量新颖的节点嵌入技术[36,37]。节点被描述为一个相对合适的低维空间中的点,这使得对多元数据应用现有的各种聚类方法成为可能。重要的是要认识到,这些相似性概念自然而然会导致不同的节点聚类。在这些选项之间的抉择必须由生物学问题而不是由它们在软件工具中的可用性来驱动(技巧1)。

技巧七:模拟网络时不要选择简单的方法
为了突出观察到的网络的特性(例如一个特殊的度量值),通常的做法是与模拟网络进行比较。用这些特性来检测是否与在模拟网络中实现的典型行为有或没有显著的偏差。然而,典型网络没有通用的定义,因此,可以检测的特征在很大程度上取决于用于模拟网络的零模型(null model)。必须为给定目的选择此零模型,来拟合预期行为,用以对比我们感兴趣的行为。换句话说,它必须合理地拟合数据以避免许多错误发现,但不能太好以至于可能将误差当成发现。
默认的选项可以包括在一系列随机图模型中选择一个零模型(例如,Erd ̈os-R ́enyi,小世界,无标度,SBM,指数随机图,配置模型(configuration model))。但是,我们建议不要太匆忙地使用它们,因为它们往往过于通用。例如,用于检测具有非常高的节点度的节点,Erd ̈os-R ́enyi模型(所有的边都是独立且等概率出现)通常是一个很差的零模型。实际上,它引起的泊松分布与大多数网络中观察到的泊松分布相差甚远,许多节点似乎意外地连接起来。相反,对于configuration model,没有任何真实网络能够包含具有度数出乎意料的高的节点,因为该零模型可以精确地调控图中每个节点的度。
此外,分析者通常关注应该由模拟网络显示出来的一系列属性:例如:节点度数的不平衡分布,与可用辅助信息相关的不同节点对应的功能,不可能出现的相互关系(例如,食物网中与体重相关的联系[38])等。这些预期的特性必须在模拟过程中被编码(例如固定度序列[34]),否则它们将出现并被检测为显著性的,或会造成检测到假的显著关系这样的副作用。例如,当评估给定转录网络中的前馈环路的数量是否是超出预期时,模拟过程必须依赖于固定数量的节点和度数,同时对允许节点中的环路的数量保持可变。
最后,当所研究的网络不是直接观测到的,而是由原始的资料解释得到,有必要模拟整个创建过程。考虑从移动数据推断的联系网络的情况[23]:可以模拟保留原始数据某些属性的运动轨迹网络,然后建立联系网络,或直接模拟“现实”的联系网络。前一种方法从本质上考虑了构建步骤引起的不确定性和偏差,而后一种方法可能会忽略这些不确定性和偏差。
技巧八:重新考虑数据以构建多个网络层
网络可以是数据聚合的结果。事实上,通常可以在不同时间、位置或条件下观察到相互作用。因此,强烈建议你现在就将不同的数据层(时间,空间,类型......)铭记于心并考虑由多个层组成的网络,因为多层网络可以提供与聚合网络相比更新的见解[39-41]。
当使用连续轮次数据收集的时间序列的网络(节点列表可能随时间变化)快照时,该网络被称为动态网络( Dynamic network )。在这种情况下,可以评估随时间变化的网络结构(例如,重新连接的交互作用或随时间变化的网络指标)以及将技巧6中提到的概念的扩展应用于动态案例中[42,43]。例如,可以从动态网络推断出动态的动物社会结构来,以增强对疾病传播的理解[44]。另一方面,在不同的空间位置也可以观察到相互作用。在生态学中,它们通常聚合成为一个元网络( Metanetwork )(或原网络( Mwtaweb ) [45])中,以研究局部网络与这种元网络的不同,并用环境因素解释这些变化。在这两种情况下,多个网络层允许将网络描述为不断演变的对象,分析旨在识别交互时空变化因素及驱动它们的因素。
在节点之间也可以观察到不同类型的交互。堆叠的层可表示不同人体组织中分子相互作用 [46]或映射突触外和突触连接体[47]组成的多重网络:在任意两个节点之间,可能存在多于一个边的情况,而每种交互类型最多一条边(通常用不同的颜色标识)。综合考虑不同的层可以增强对节点相互作用的理解。例如,与使用单层相比,联合使用营养和非营养交互作用可以增强对物种生态角色的定义[34]。最后,还可以将不同层的信息与每一层的不同类型的节点集集成在一起,例如蛋白质和化合物[48]。在这种情况下,在层内和层间定义了不同类型的交互作用。在所有这些情况下,不同的信息层被整合到全面的网络中,使得它们被联合处理而不是一个接一个地处理。
技巧九:跨越你的学科,研读网络科学文献
网络科学是在21世纪初出现的新兴学科。现在已经出现在来自不同领域的如物理学,统计学,计算机科学或社会科学的研究人员的超活跃社区。已经存在了大量关于网络的文献,生物学家想要深入研究它是一项挑战。确实,我们并不习惯在我们的研究领域之外探索参考书目。参考文献[5,41,49,50]和评论[22,39,51]显然是提升网络技能的良好切入点。但是毫无疑问,如果你努力学习和你研究学科中不同的专业术语,那么你将从网络科学领域的文献中受益匪浅。
结论
这里介绍的9个技巧提供了数据分析者入门网络数据分析的一种方式。这些技巧并不是唯一的,我们知道其他一些基于网络的问题例如网络中信息的传播也值得特别关注。尽管如此,非专业网络分析人员必须对自己逐步学习网络概念和方法的能力充满信心,网络的概念和方法会对他们的科学研究产生正面的影响。
原文题目:
Nine Quick Tips for Analyzing Network Data
原文地址:
参考文献:
1. T. Ideker and R. Nussinov, “Network approaches and applications in biology,”PLoS computational biology, vol. 13, no. 10, p. e1005771, 2017.
2. M. Zitnik, R. Sosi, and J. Leskovec, “Prioritizing network communities,” NatCommun, vol. 9, no. 1, p. 2544, 2018.
3. B. Wang, A. Pourshafeie, M. Zitnik, J. Zhu, C. D. Bustamante, S. Batzoglou, andJ. Leskovec, “Network enhancement as a general method to denoise weightedbiological networks,” Nat Commun, vol. 9, no. 1, p. 3108, 2018.
4. R. E. Kass, B. S. Caffo, M. Davidian, X.-L. Meng, B. Yu, and N. Reid, “Tensimple rules for effective statistical practice,” 2016.
5. R. Diestel, “Graph theory.” http://diestel-graph-, 2016.
6. A. Barrat, M. Barthelemy, R. Pastor-Satorras, and A. Vespignani, “Thearchitecture of complex weighted networks,” Proceedings of the national academyof sciences, vol. 101, no. 11, pp. 3747–3752, 2004.
7. P. P. Staniczenko, J. C. Kopp, and S. Allesina, “The ghost of nestedness inecological networks,” Nature communications, vol. 4, p. 1391, 2013.
8. G. A. Pavlopoulos, P. I. Kontou, A. Pavlopoulou, C. Bouyioukos, E. Markou, andP. G. Bagos, “Bipartite graphs in systems biology and medicine: a survey ofmethods and applications,” GigaScience, vol. 7, no. 4, p. giy014, 2018.
9. V. Miele, F. Picard, and S. Dray, “Spatially constrained clustering of ecologicalnetworks,” Methods in Ecology and Evolution, vol. 5, no. 8, pp. 771–779, 2014.
10. P. Expert, T. S. Evans, V. D. Blondel, and R. Lambiotte, “Uncoveringspace-independent communities in spatial networks,” Proceedings of the NationalAcademy of Sciences, vol. 108, no. 19, pp. 7663–7668, 2011.
11. M. Mariadassou, S. Robin, C. Vacher, et al., “Uncovering latent structure invalued graphs: a variational approach,” The Annals of Applied Statistics, vol. 4,no. 2, pp. 715–742, 2010.
12. M. A. Fortuna, R. G. Albaladejo, L. Fern´andez, A. Aparicio, and J. Bascompte,“Networks of spatial genetic variation across species,” Proceedings of the NationalAcademy of Sciences, vol. 106, no. 45, pp. 19044–19049, 2009.
13. D. Hric, R. K. Darst, and S. Fortunato, “Community detection in networks:Structural communities versus ground truth,” Physical Review E, vol. 90, no. 6,p. 062805, 2014.
14. R. Rossi and N. Ahmed, “The network data repository with interactive graphanalytics and visualization,” in Twenty-Ninth AAAI Conference on ArtificialIntelligence, 2015.
15. G. K. Sandve, A. Nekrutenko, J. Taylor, and E. Hovig, “Ten simple rules forreproducible computational research,” PLoS Comput. Biol., vol. 9, no. 10,p. e1003285, 2013.
16. M. A. Carey and J. A. Papin, “Ten simple rules for biologists learning toprogram,” PLoS Comput. Biol., vol. 14, no. 1, p. e1005871, 2018.
17. J. W. Tukey, Exploratory data analysis. Reading: Addison-Wesley, 1977.
18. M. Krzywinski, I. Birol, S. J. Jones, and M. A. Marra, “Hive plots—rationalapproach to visualizing networks,” Briefings in Bioinformatics, vol. 13, no. 5,pp. 627–644, 2011.
19. M. Rubinov and O. Sporns, “Complex network measures of brain connectivity:uses and interpretations,” Neuroimage, vol. 52, no. 3, pp. 1059–1069, 2010.
20. J. Bascompte, P. Jordano, C. J. Meli´an, and J. M. Olesen, “The nested assemblyof plant–animal mutualistic networks,” Proceedings of the National Academy ofSciences, vol. 100, no. 16, pp. 9383–9387, 2003.
21. R. E. Van Dijk, J. C. Kaden, A. Arg¨uelles-Tic´o, D. A. Dawson, T. Burke, andB. J. Hatchwell, “Cooperative investment in public goods is kin directed incommunal nests of social birds,” Ecology letters, vol. 17, no. 9, pp. 1141–1148,2014.
22. S. Fortunato and D. Hric, “Community detection in networks: A user guide,”Physics Reports, vol. 659, pp. 1–44, 2016.
23. D. R. Farine and H. Whitehead, “Constructing, conducting and interpretinganimal social network analysis,” J Anim Ecol, vol. 84, no. 5, pp. 1144–1163, 2015.
24. A. Costa, A. M. M. Gonzalez, K. Guizien, A. M. Doglioli, J. M. Gomez,A. Petrenko, and S. Allesina, “Ecological networks: Pursuing the shortest path,however narrow and crooked,” bioRxiv, p. 475715, 2018.
25. B. C. Van Wijk, C. J. Stam, and A. Daffertshofer, “Comparing brain networks ofdifferent size and connectivity density using graph theory,” PloS one, vol. 5,no. 10, p. e13701, 2010.
26. L. Pellissier, C. Albouy, J. Bascompte, N. Farwig, C. Graham, M. Loreau, M. A.Maglianesi, C. J. Meli´an, C. Pitteloud, T. Roslin, et al., “Comparing speciesinteraction networks along environmental gradients,” Biological Reviews, vol. 93,no. 2, pp. 785–800, 2018.
27. S. Fortunato and M. Barthelemy, “Resolution limit in community detection,”Proceedings of the National Academy of Sciences, vol. 104, no. 1, pp. 36–41, 2007.
28. S. Kortsch, R. Primicerio, M. Aschan, S. Lind, A. V. Dolgov, and B. Planque,“Food-web structure varies along environmental gradients in a high-latitudemarine ecosystem,” Ecography, vol. 42, no. 2, pp. 295–308, 2019.
29. H. K. Norton, D. J. Emerson, H. Huang, J. Kim, K. R. Titus, S. Gu, D. S.Bassett, and J. E. Phillips-Cremins, “Detecting hierarchical genome folding withnetwork modularity,” Nature methods, vol. 15, no. 2, p. 119, 2018.
30. S. Allesina and M. Pascual, “Food web models: a plea for groups,” Ecol. Lett.,vol. 12, no. 7, pp. 652–662, 2009.
31. M. E. Newman and E. A. Leicht, “Mixture models and exploratory analysis innetworks,” Proceedings of the National Academy of Sciences, vol. 104, no. 23,pp. 9564–9569, 2007.
32. R. F. Betzel, J. D. Medaglia, and D. S. Bassett, “Diversity of meso-scalearchitecture in human and non-human connectomes,” Nature Communications,vol. 9, no. 1, p. 346, 2018.
33. J.-J. Daudin, F. Picard, and S. Robin, “A mixture model for random graphs,”Statistics and computing, vol. 18, no. 2, pp. 173–183, 2008.
34. S. K´efi, V. Miele, E. A. Wieters, S. A. Navarrete, and E. L. Berlow, “HowStructured Is the Entangled Bank? The Surprisingly Simple Organization ofMultiplex Ecological Networks Leads to Increased Persistence and Resilience,”PLoS Biol., vol. 14, no. 8, p. e1002527, 2016.
35. D. B. Stouffer, M. Sales-Pardo, M. I. Sirer, and J. Bascompte, “Evolutionaryconservation of species’ roles in food webs,” Science, vol. 335, no. 6075,pp. 1489–1492, 2012.
36. B. Perozzi, R. Al-Rfou, and S. Skiena, “Deepwalk: Online learning of socialrepresentations,” in Proceedings of the 20th ACM SIGKDD InternationalConference on Knowledge Discovery and Data Mining, KDD ’14, (New York, NY,USA), pp. 701–710, ACM, 2014.
37. A. Grover and J. Leskovec, “node2vec: Scalable feature learning for networks,” inProceedings of the 22nd ACM SIGKDD international conference on Knowledgediscovery and data mining, pp. 855–864, ACM, 2016.
38. U. Brose, T. Jonsson, E. L. Berlow, P. Warren, C. Banasek-Richter, L.-F. Bersier,J. L. Blanchard, T. Brey, S. R. Carpenter, M.-F. C. Blandenier, et al.,“Consumer–resource body-size relationships in natural food webs,” Ecology,vol. 87, no. 10, pp. 2411–2417, 2006.
39. S. Boccaletti, G. Bianconi, R. Criado, C. I. Del Genio, J. G´omez-Gardenes,M. Romance, I. Sendina-Nadal, Z. Wang, and M. Zanin, “The structure anddynamics of multilayer networks,” Physics Reports, vol. 544, no. 1, pp. 1–122,2014.
40. S. Pilosof, M. A. Porter, M. Pascual, and S. K´efi, “The multilayer nature ofecological networks,” Nature Ecology & Evolution, vol. 1, no. 4, p. 0101, 2017.
41. G. Bianconi, Multilayer Networks: Structure and Function. Oxford universitypress, 2018.
42. C. Matias and V. Miele, “Statistical clustering of temporal networks through adynamic stochastic block model,” Journal of the Royal Statistical Society: SeriesB (Statistical Methodology), vol. 79, no. 4, pp. 1119–1141, 2017.
43. G. Rossetti and R. Cazabet, “Community discovery in dynamic networks: asurvey,” ACM Computing Surveys (CSUR), vol. 51, no. 2, p. 35, 2018.
44. D. Farine, “The dynamics of transmission and the dynamics of networks,”Journal of Animal Ecology, vol. 86, no. 3, pp. 415–418, 2017.
45. M. Ohlmann, V. Miele, S. Dray, L. Chalmandrier, L. O’Connor, and W. Thuiller,“Diversity indices for ecological networks: a unifying framework using Hillnumbers,” Ecology letters, 2019.
46. M. Zitnik and J. Leskovec, “Predicting multicellular function through multi-layertissue networks,” Bioinformatics, vol. 33, no. 14, pp. i190–i198, 2017.
47. B. Bentley, R. Branicky, C. L. Barnes, Y. L. Chew, E. Yemini, E. T. Bullmore,P. E. V´ertes, and W. R. Schafer, “The multilayer connectome of caenorhabditiselegans,” PLoS computational biology, vol. 12, no. 12, p. e1005283, 2016.
48. A. J. Berenstein, M. P. Magari˜nos, A. Chernomoretz, and F. Ag¨uero, “Amultilayer network approach for guiding drug repositioning in neglected diseases,”PLoS neglected tropical diseases, vol. 10, no. 1, p. e0004300, 2016.
49. A.-L. Barab´asi, “Network science.” , 2019.
50. M. Newman, Networks. Oxford university press, 2018.
51. P. Goyal and E. Ferrara, “Graph embedding techniques, applications, andperformance: A survey,” Knowledge-Based Systems, vol. 151, pp. 78–94, 2018.
作者:Vincent Miele , Stéphane Dray , Catherine Matias , Stéphane Robin
翻译:王梓任
审校:郭瑞东
编辑:张爽
话题:
0
推荐
财新博客版权声明:财新博客所发布文章及图片之版权属博主本人及/或相关权利人所有,未经博主及/或相关权利人单独授权,任何网站、平面媒体不得予以转载。财新网对相关媒体的网站信息内容转载授权并不包括财新博客的文章及图片。博客文章均为作者个人观点,不代表财新网的立场和观点。




 京公网安备 11010502034662号
京公网安备 11010502034662号 